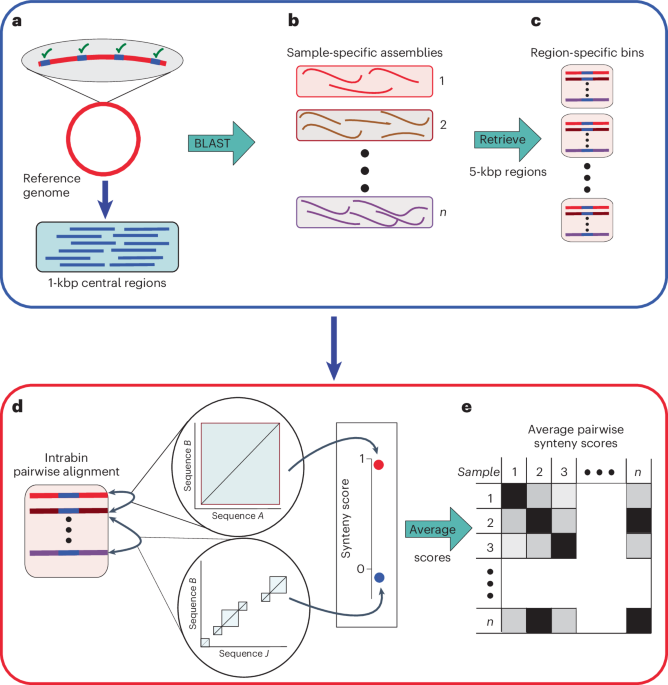
SynTracker 管道
SynTracker 流程由三个主要部分组成。在第一部分中,SynTracker 接受一组参考基因组(每个物种一个基因组),可以是完全组装的,也可以是重叠群的集合。每个物种的参考被分割成一组 1 kbp 中心区域,这些区域被装箱并存储在一起。
在第二部分中,SynTracker 根据用户提供的宏基因组组装或基因组集合创建一个 BLAST 数据库。接下来,它针对新建的 BLAST 数据库对每个中心区域执行 BLAST 搜索,最低同一性为 97%,最低查询覆盖率为 70%(即 700 bp)。在此部分的最后一步中,除了使用“blastcmddb”命令检索 BLAST 搜索的匹配项外,还检索 BLAST 匹配项两侧的 2 kbp 区域。匹配项上游和下游 <2 kbp 范围内的匹配项将被排除在进一步分析之外。每个检索到的序列都由其来源样本和参考基因组中的匹配区域表示。
在流程的第三部分中,基因组片段按其在参考基因组中的匹配区域分组,并进行成对比对以识别每对序列中的同源块。使用 DECIPHER R 包中的“FindSynteny”函数来识别每对比对中的同源块21,参数 'maxGap' 和 'maxSep' 均设置为 15。此外,下游分析仅考虑重叠度最小为 4,800 bp 的成对比较。接下来,根据每个成对比对计算连锁得分:
$${rm{synScore}}={1}+{{{log }}}_{{10}}left(left({rm{Ov}/{len}}right)/{B}right)$$
其中 Ov 代表成对比对中识别出的重叠连锁块的累积长度,len 表示每对中较短序列的长度, 乙 代表每次成对比对中识别的连锁块的数量。
在流程第三部分的最后一步,对于每个参考基因组, n 每对宏基因组样本或基因组随机选择基因组区域。APSS 是通过平均个体成对同源性得分来计算的。样本或基因组对少于 n 区域被排除在下游分析之外。
计算机进化模拟
如上所述计算每组采样细胞的共线性得分。然而,由于模拟中使用的基因组片段长度限制为 20 kbp,共线性得分基于每对模拟基因组的 ~20 kbp 区域的单次比对。通过应用 Welch 统计分析,对基于 indel 和基于 SNP 的模拟中 BLAST 身份或共线性得分之间的差异进行统计显著性分析 吨– 在上述指定的时间点对整组成对比较进行检验。
细菌基因组的分类
计算 APSS 值 埃。 大肠杆菌 菌株对如上所述进行,并使用 埃。大肠杆菌 str. K-12 亚株 MG1655 基因组作为参考(美国国家生物技术信息中心 (NCBI) 参考序列: NC_000913.3)。
使用以下方法构建基于连锁的系统发育树 n 每对比较的区域( n= 20–200)。对于每个成对比较的区域数,APSS 值被转换为同源距离,其等于 APSS − 1 的绝对值。所有成对同源距离都放置在对称矩阵中,该矩阵用于使用 R 包 'phangorn' 计算非加权成对组法和算术平均系统发育树51。
从文献中导入了完整的基于 Mash 距离的系统发育树,其中包括 10,000 多个基因组24 ('Supplementary_Data_Fig_3c_10k_newick.nwk' 文件)。树标签从 MD5 转换为组装接入,然后修剪基于 Mash 的树,以便仅显示与 140 埃 。 大肠杆菌用 SynTracker 分析的基因组被保留。
通过使用“TreeDist”R 包计算基于信息的广义 RFD,确定基于连锁的系统发育树和基于 Mash 的树之间的距离52。
通过打乱基于连锁的树的标签(每次比较 100,000 次迭代)并计算随机树中 RFD 等于或低于实际基于连锁的树和基于 Mash 的树之间距离的比例,来计算每次树比较的显著性水平。
实验进化的应变比较 埃 。 大肠杆菌人口
菌株比较 H 。 幽门螺杆菌临床分离株, 否 。 淋病杆菌分离和 年代 。 有趣的分离株
共77个临床 H 。 幽门螺杆菌分离株2,20个图书馆 年代 。 有趣的分离株和 12 个来自临床分离株的文库 否 。 淋病杆菌取自 NCBI SRA 数据库(补充表 3 和 5),按照上述方法进行质量过滤和组装。对于 inStrain 和 SynTracker 分析,使用相同的参考基因组: H。 幽门螺杆菌,GenBank:CP032479.1;年代。 有趣的, GCF_000331185.2_ASM33118v2; 否。淋病杆菌, GCF_900087635.2在 SynTracker 分析中,每对比较使用 200 个区域,但否。淋病杆菌,其中每对比较使用 60 个区域。
全基因组比对H。幽门螺杆菌 。淋病杆菌和年代 。有趣的 程序集
使用 Mauve 软件进行全基因组比对55.首先,使用上面描述的参考基因组对每个组装的重叠群进行排序,然后使用软件的默认参数相互对齐。
基于 SNP 和连锁的人类肠道宏基因组分析
对于综合分析,我们进一步利用了最近发表的 Strain 和 SynTracker 对婴儿-成人对的分析三十四。为了创建 APSS 与 popANI 图,我们使用了两种工具识别的菌株比较子集(>12,000 个菌株对)。SynTracker 分析使用每对比较 30 个区域进行。
为了识别具有优先种内基因组多样性模式的物种,我们首先根据 inStrain(使用 popANI 作为距离矩阵)和 SynTracker(APSS)计算每个物种在 5% 最相似菌株对中的富集度。使用超几何分布概率检验确定每个物种的富集度:
$${p}({X}={k})=(({K}/k)(({N}-{K})/(nk)))/({N}/n)$$
在哪里否 是每个数据集中成对比较的次数,n是最相似的成对比较的数量(即 5%否 ),钾 是整个数据集中每个物种的成对比较次数,钾 是最相似子集中每个物种的成对比较的次数。
确定 SynTracker 的性能
对于所研究的 33 个物种,我们使用了公开的参考基因组(补充表 9),如上所述,该基因组被分割成 1 kbp 中心区域的集合。接下来,我们执行了每个样本的从头宏基因组组装,以构建我们的搜索空间 (方法 和图。 1)。宏基因组组装体被随机分为训练集和测试集(117 个和 106 个样本,分别来自 45 名和 43 名供体)。对于这两个集合,我们在随机选择后为每个物种计算了八个不同的最终 APSS 矩阵n 每对比较的区域(n= 15–200 个 5 kbp 区域)。然后,我们将训练集中的成对比较分为来自同一宿主的不同时间点(宿主内组)和来自不同宿主(宿主间组)。以训练集中成对比较的分类为基本事实,我们生成了 ROC56 确定不同 APSS 值下分类的特异性和敏感性。我们根据每个物种和子采样值组合的 100 次迭代绘制曲线。
接下来,我们的目标是使用 33 种物种测试集评估 SynTracker 的性能。我们首先确定了能够最佳区分宿主内比较和宿主间比较的 APSS 值。APSS 高于这些“最佳”值的成对比较被视为同株比较,而低于这些值的成对比较被视为不同菌株。这是通过计算J指数31 对于每个物种和子采样深度的组合。我们将每个采样级别的“阈值 APSS”定义为每个成对比较中给定数量的基因组区域的所有物种的最佳 APSS 的平均值。最后,我们通过将这些 APSS 阈值引入测试集来确定我们方法的特异性和敏感性。为了避免抽样偏差,我们对每个物种和采样级别的组合重复了 100 次此过程,并计算了平均特异性和敏感性值。正如预期的那样,我们观察到子采样区域的数量与敏感性和特异性之间存在直接对应关系(扩展数据图)。 7 和补充表 8),使用每对比较 200 个区域计算得出的最大平均灵敏度和特异性值为 98.6% 和 97%。虽然每对比较的区域数量较少通常会导致准确度较低,但使用此类值的决定可能是通过在分析中包含更多样本来证明的。研究人员可以决定是否优先提高准确度或样本量。
对 SynTracker 进行基准测试
为了对 SynTracker 进行基准测试,我们首先下载了作者执行测试时使用的相同原始读取文件二十八. 质量过滤和从头组装如上所述进行。为了与其他工具进行更准确的比较,我们使用了作者组装的 SRG(即代表 ANI > 99% 的 MAG 簇的基因组)二十八 作为SynTracker的参考基因组输入。ROC图的准备如上所述。
母婴病毒传播
从 NCBI SRA 数据库下载宏基因组样本,并按上述方法进行质量过滤和组装。但是,由于某些样本的两个匹配读取文件中只有一个通过了我们的质量过滤,因此我们使用单端读取进行宏基因组组装。
不同地理位置的菌株相似性
为了避免母婴菌株传播对区域内连锁得分的影响,我们过滤掉了母亲和自己婴儿之间的菌株比较。我们还删除了不是出生在他们目前居住的国家的受试者,以消除当地人口和外籍人士之间的比较。受试者的位置基于每个受试者提供的居住地。
省内和省间菌株比较差异的统计学意义由 Wilcoxon–Mann–Whitney 检验确定,并使用 Benjamini–Hochberg 校正对检验结果进行多重检验校正,如上所述。匹配效应大小(Cohen'sd) 使用“effsize”R 包(v.0.8.1)进行计算和分类58. 使用 CLANS 软件生成物种特定网络59,其中 −log10(APSS)用作边权重。分析中要考虑的最小 APSS 分数为 ~0.75。从网络图中删除了单身人士(即网络中没有连接的主体)。
参考基因组的推荐使用
由于最先进的宏基因组基因组组装方法产生了越来越多的 MAG 集合,我们建议遵循 inStrain 程序工作流程二十八,其中 MAG 集合被聚类以创建 SRG,用作我们流程中的参考基因组。这种方法应该可以扩展新物种的研究菌株。
报告摘要
2024-06-19 00:00:00
1718874468