Doudna,JA 治疗性基因组编辑的前景与挑战。 自然 578229–236 (2020年)。
Raguram, A.、Banskota, S. 和 Liu, DR 基因编辑剂的治疗性体内递送。 细胞 185,2806–2827(2022年)。
Wang, D., Zhang, F. 和 Gao, G. 基于 CRISPR 的治疗性基因组编辑:策略和通过 AAV 载体的体内递送。 细胞 181136–150 (2020年)。
Kim, D., Luk, K., Wolfe, SA 和 Kim, J.-S. 评估和增强基因编辑核酸酶和脱氨酶的靶标特异性。 生物化学年鉴。 88191–220 (2019)。
Tao, J.、Bauer, DE 和 Chiarle, R. 评估和提高 CRISPR-Cas 工具的安全性:从 DNA 到 RNA 编辑。 纳特。 常见的。 14,212 (2023)。
Kim, D. 等人 Digenome-seq:人类细胞中 CRISPR–Cas9 脱靶效应的全基因组分析。 天然方法 12,237–243 (2015)。
Cameron, P. 等人。绘制 CRISPR–Cas9 切割的基因组图谱。 天然方法 14,600–606 (2017年)。
Tsai, SQ 等人 CIRCLE-seq:一种针对全基因组 CRISPR–Cas9 核酸酶脱靶的高灵敏度体外筛选方法。 天然方法 14,607–614 (2017年)。
Tsai, SQ 等人 GUIDE-seq 能够对 CRISPR–Cas 核酸酶的脱靶切割进行全基因组分析。 天然生物技术。 33,187–197 (2015)。
Wienert, B. 等人使用 DISCOVER-Seq 在体内无偏检测 CRISPR 脱靶。 科学 364286–289 (2019)。
Zou, RS 等人利用 DISCOVER-Seq+ 提高体内 CRISPR 脱靶检测的灵敏度。 天然方法 20,706–713(2023年)。
Urnov, FD、Rebar, EJ、Holmes, MC、Zhang, HS 和 Gregory, PD 利用工程化锌指核酸酶进行基因组编辑。 夜晚。 牧师。 基因。 11,636–646 (2010)。
Joung, JK 和 Sander, JD TALENs:一种广泛适用的靶向基因组编辑技术。 Nat. Rev. Mol. Cell Biol. 1449–55 (2013)。
Cong, L. 等人利用 CRISPR/Cas 系统进行多重基因组工程。 科学 339,819–823 (2013)。
Anzalone, AV、Koblan, LW 和 Liu, DR 使用 CRISPR-Cas 核酸酶、碱基编辑器、转座酶和引发编辑器进行基因组编辑。 天然生物技术。 三十八824–844 (2020年)。
Komor, AC、Kim, YB、Packer, MS、Zuris, JA 和 Liu, DR 无需双链 DNA 切割即可对基因组 DNA 中的目标碱基进行可编程编辑。 自然 533,420–424 (2016)。
Anzalone, AV 等人。无需双链断裂或供体 DNA 的搜索和替换基因组编辑。 自然 576149–157 (2019)。
Gaudelli, NM 等人。无需 DNA 切割即可对基因组 DNA 中的 A•T 进行可编程碱基编辑至 G•C。 自然 551,464–471 (2017)。
Liu, T. & Huang, J. 复制蛋白 A 及更多:真核细胞中的单链 DNA 结合蛋白。 Acta Biochim Biophys. Sin. 四十八,665–670 (2016)。
Wold,MS复制蛋白A:一种异三聚体、单链DNA结合蛋白,是真核生物DNA代谢所必需的。 生物化学年鉴。 66,61–92 (1997)。
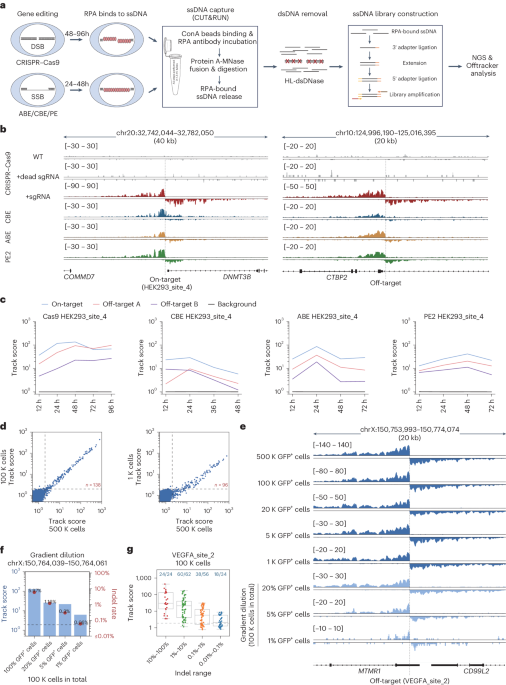
Skene, PJ, Henikoff, JG 和 Henikoff, S. 针对低细胞数量进行高效的原位全基因组分析。 天然产物研究方法 十三1006–1019 (2018)。
Kabeche, L., Nguyen, H., Buisson, R. 和 Zou, L. 有丝分裂特异性和 R 环驱动的 ATR 通路促进忠实的染色体分离。 科学 359108–114 (2018)。
Gan, X. 等人。适当的 RPA 乙酰化可促进准确的 DNA 复制和修复。 核酸研究。 51,5565–5583(2023年)。
Amemiya, HM, Kundaje, A. 和 Boyle, AP ENCODE 黑名单:识别基因组中的问题区域。 科学报告 9,9354(2019年)。
Ferrari, M.、Twayana, S.、Marini, F. 和 Pellicioli, A. In 基因组不稳定性:方法和方案 (Muzi-Falconi,M. & Brown,GW 编辑)119–129(Humana Press,2017 年)。
Stoler, N. 和 Nekrutenko, A. Illumina 测序仪器的测序错误概况。 纳尔通过。 生物信息。 3,lqab019(2021)。
Kim, D., Kim, S., Kim, S., Park, J. 和 Kim, J.-S. 多重 Digenome-seq 揭示的 CRISPR–Cas9 核酸酶的全基因组靶标特异性。 基因组研究。 二十六,406–415 (2016)。
文章
中科院
PubMed
PubMed Central
谷歌学术
Lei, Z. 等人 Detect-seq 揭示了胞嘧啶碱基编辑器进行的原间隔区外编辑和靶链编辑。 天然方法 18,643–651(2021年)。
Liang, P. 等人通过 EndoV-seq 对腺嘌呤碱基编辑器特异性进行全基因组分析。 纳特。 常见的。 10,67(2019)。
Liang, S.-Q. 等人使用 PE 标签对体外和体内的 Prime 编辑器脱靶位点进行全基因组分析。 天然方法 20,898–907(2023年)。
Kim, DY、Moon, SB、Ko, J.-H.、Kim, Y.-S. 和 Kim, D. 对人类细胞中主要编辑系统特异性的公正调查。 核酸研究。 四十八,10576–10589(2020年)。
Zuo, E. 等人胞嘧啶碱基编辑器在小鼠胚胎中产生大量脱靶单核苷酸变体。 科学 364289–292 (2019)。
Duan, J. 等人。人类基因组中 CRISPR/Cas9 脱靶的全基因组识别。 细胞研究。 24,1009–1012(2014年)。
Frangoul, H. 等人。CRISPR–Cas9 基因编辑用于治疗镰状细胞病和β-地中海贫血。 N. 英格兰。 J. Med。 384252–260 (2021)。
Nakamura-Ishizu, A., Takizawa, H. 和 Suda, T. 造血干细胞静止的分析、作用和调节。 发展 141,4656–4666 (2014年)。
Chang, HHY、Pannunzio, NR、Adachi, N. 和 Lieber, MR 非同源 DNA 末端连接和双链断裂修复的替代途径。 Nat. Rev. Mol. Cell Biol. 18495–506 (2017)。
Wienert, B.、Wyman, SK、Yeh, CD、Conklin, BR 和 Corn, JE 使用 DISCOVER-seq 进行 CRISPR 脱靶检测。 天然产物研究方法 15,1775–1799(2020年)。
Wheeler, DL 等人。美国国家生物技术信息中心的数据库资源。 核酸研究。 33,D39–D45(2005)。
Zhang, H. 等. 使用 Chromap 对染色质谱进行快速比对和预处理。 纳特。 常见的。 12,6566(2021年)。
Quinlan, AR 和 Hall, IM BEDTools:一套用于比较基因组特征的灵活实用程序。 生物信息学 二十六,841–842 (2010)。
Kent, WJ、Zweig, AS、Barber, G.、Hinrichs, AS 和 Karolchik, D. BigWig 和 BigBed:支持浏览大型分布式数据集。 生物信息学 二十六2204–2207 (2010年)。
Ramirez, F. 等人 deepTools2:用于深度测序数据分析的下一代网络服务器。 核酸研究。 四十四,W160–W165(2016年)。
Langmead, B. & Salzberg, SL 使用 Bowtie 2 进行快速间隙读取比对。 天然方法 9,357–359 (2012)。
Danecek, P. 等人。SAMtools 和 BCFtools 的十二年历程。 超级科学 10,giab008(2021年)。
Bleckwehl, T. 等人。增强子相关的 H3K4 甲基化保障体外生殖系能力。 纳特。 常见的。 12,5771(2021年)。
Man, N. 等人,p300 抑制骨髓增生异常综合征向急性髓细胞白血病的转变。 JCI 洞察 6,e138478 (2021)。
Ji, L. et al. TOPORS 是一种肿瘤抑制蛋白,有助于维持高阶染色质结构。 生物化学与生物物理学报基因调控机械学报。 1863,194518(2020年)。
Lex, RK 等人 GLI 转录抑制调节响应 Hedgehog 信号传导的组织特异性增强子活性。 埃利夫 9,e50670(2020年)。
Boix, CA、James, BT、Park, YP、Meuleman, W. 和 Kellis, M. 通过整合表观基因组学研究人类疾病基因座的调控基因组回路。 自然 590,300–307 (2021)。
Clement, K. 等人 CRISPResso2 提供准确、快速的基因组编辑序列分析。 天然生物技术。 三十七224–226 (2019)。
Zhu, M. et al。 Tracking-seq 揭示了 CRISPR/Cas9 介导的基因组编辑中脱靶效应的异质性。 美国国立生物技术信息中心 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE236360 (2024)。
徐,R. Offtracker。 GitHub https://github.com/Lan-lab/offtracker (2024)。
2024-07-02 00:00:00
1720163845





/https://i.s3.glbimg.com/v1/AUTH_bc8228b6673f488aa253bbcb03c80ec5/internal_photos/bs/2024/Y/K/l6x8MSQKyB0jODe9fmSw/vic-1160.jpg)